脑图像分析基本环境配置:MATLAB + SNAP + DiffusionKit + SPM12 + CAT12
日期: 2019-05-19 分类: 跨站数据测试 761次阅读
1、安装MATLAB,此处版本为MATLAB R2015b。
2、使用ADNI作为实验数据集。安装SNAP用于查看.nii图像,此处版本为ITK-SNAP 3.8。
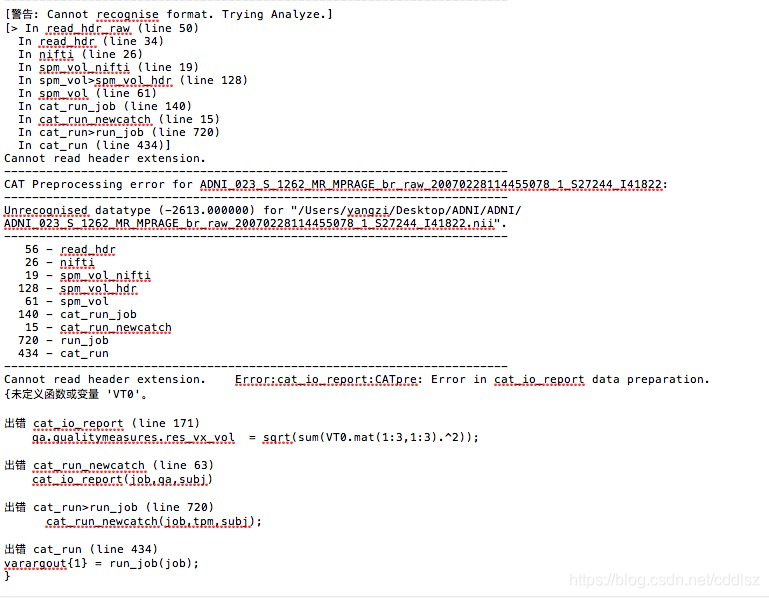
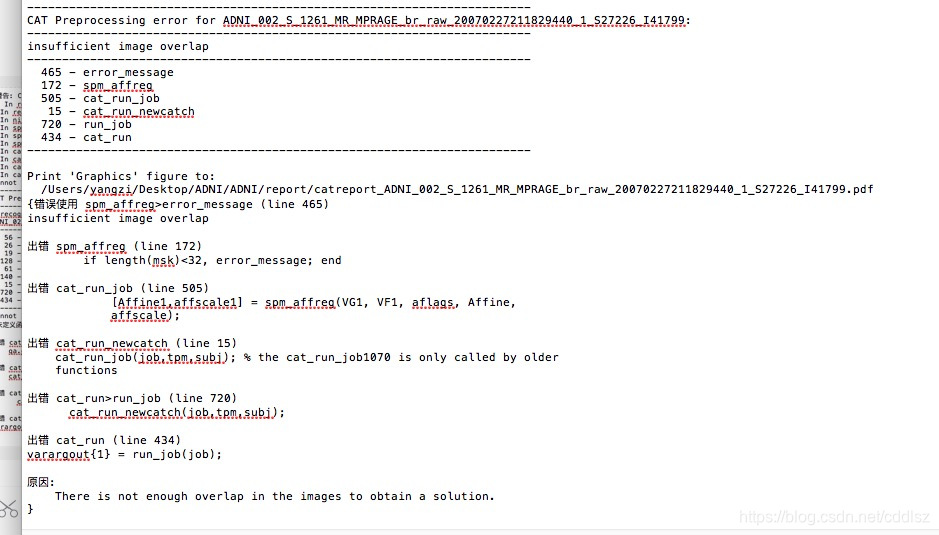
3、使用DiffusionKit进行图像配准。若未配准,后期可能会出现以下错误:
配准方法:
(1)在Brainnetome官网下载DiffusionKit工具包并安装(点击此处查看使用指南)。
(2)打开CMD进入DiffusionKit目录,通过如下指令查看reg_aladin.exe使用方式:
reg_aladin.exe –h可以看到使用方法为:

其中,-ref <filename>是指标准模板,-flo <filename>是指源图像。
(3)OK,下载一个标准模板,这里使用MNI_152。
(4)使用如下指令进行图像配准:
![]()
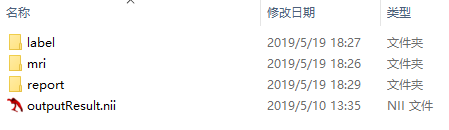
这样就生成了配准后的文件:outputResult.nii。
4、使用SPM12软件包进行脑图像数据分析。
(1)下载并安装SPM12。
(2)打开MATLAB,在菜单栏中点击“设置路径”->“添加并包含子文件夹”,找到spm12的根目录添加即可。
5、使用CAT12进行脑图像分割。CAT是SPM的计算解剖工具箱,具有与VBM相似的功能。
(1)下载并安装CAT12。
(2)由于是要在SPM中使用CAT,因此要把cat12放在\spm12\toolbox\目录下。
6、到此为止,基本环境已经配置完毕,接下来以一个nii图像的分割为例进行CAT12使用方法的演示。
(1)在MATLAB命令行窗口中输入spm,打开spm12窗口。
(2)在Toolbox下拉框中选择cat12,打开工作窗口。
(3)点击左上角的Segment Data,会弹出Batch Editor窗口。
这里使用刚刚配准过的outputResult.nii作为实验数据。创建一个名为test的文件夹,放入实验数据。通常要根据数据特征修改“Affine Regularisation(仿射正则化)”参数,由于该数据来源于ADNI数据集,这一参数不需要修改。将“Voxel size for normalized images(标准化图像的体素大小)”这一参数从1.5修改为1。
(4)双击“Volumes”选项,找到test文件夹,选择实验数据,点击“Done”。
(5)这时菜单栏上的三角符号已经变亮,点击即开始进行图像分割。
(6)MATLAB会实时输出运行状态信息。

(7)经过约半小时的时间,图像已经分割完毕,输出信息保存在test文件夹下。
(8)输出展示。
①原图像 outputResult.nii
②分割后图像
③catreportj_outputResult.jpg
除特别声明,本站所有文章均为原创,如需转载请以超级链接形式注明出处:SmartCat's Blog
精华推荐